LA PRUEBA DE PATERNIDAD CON ADN
Una descripción para abogados, laboratorios clínicos y público en general
Dr. en C. Héctor Rangel Villalobos [1] |

Versión para imprimir |
Resumen
La facilidad para obtener perfiles genéticos ha revolucionado la identificación humana para resolver casos criminales así como determinar relaciones biológicas de parentesco, comúnmente paternidad. Las repeticiones cortas en tándem, más bien conocido como STRs son los marcadores genéticos más utilizados con este fin. Un perfil de ADN se compone de los genotipos para varios STRs, que forman un código casi único para diferenciar o relacionar a una persona biológicamente. Actualmente el análisis de laboratorio se basa en tres técnicas: 1) extracción de ADN, 2) PCR multiplex, y 3) electroforesis capilar, y existen kits comerciales para desarrollar fácilmente esta tarea. En paternidad se espera que entre el perfil de ADN de un supuesto padre (SP) e hijo (H) exista al menos una concordancia por cada marcador. Cuando para más de un marcador no hay concordancia entre SP y H, se establece una exclusión y prácticamente el resultado es incuestionable. Sin embargo, cuando todo concuerda entre SP y H se debe hacer una valoración bioestadística del caso, estimando la probabilidad de que algún otro individuo tomado al azar de la población pudiera concordar con el hijo. Para esto se debe haber estudiado a los marcadores STRs en la población donde se realizan las pruebas de ADN, para usar las frecuencias alélicas de los STRs en el análisis bioestadístico. A dicha estimación se le denomina índice de paternidad (IP), que indica cuantas veces es más probable haber encontrado la concordancia SP-H considerando que sí es el padre, respecto a que fuera un individuo tomado al azar de la población. Este IP puede ser transformado a porcentaje de paternidad (W), para facilitar la interpretación. Por ejemplo, un IP de 1000 se convierte en W= 99.9%. Existen formulas especificas para calcular el IP, las cuales cambian según el genotipo de cada marcador analizado, y según el caso de paternidad, ya sea que participe o no la madre, si no está el padre pero sí están los abuelos, si se duda de la paternidad de ambos padres, etc. En México existen estos estudios que sustentan su correcta aplicación, suele ser más preocupante que no hay suficiente personal capacitado, tanto en el ámbito laboratorial como en la impartición de justicia, que garanticen siempre la correcta interpretación de la prueba sobre bases científicas.
Introducción
En 1985 Alec Jeffreys implementó el uso del material genético (ADN[2]) para identificación humana, obteniendo un patrón de bandas parecido a un código de barras al que denominó huella digital del ADN (DNA fingerprinting) (Figura 1). Actualmente a esta prueba se le conoce como perfil de ADN, huella genética, o simplemente prueba de ADN. Este perfil de ADN se ha demostrado que es prácticamente único e irrepetible, a excepción de los gemelos monocigotos[3], lo que permite diferenciar a cualquier persona de otra y establecer sus relaciones biológicas de parentesco. Las aplicaciones de la prueba de ADN son diversas, pero destacan dos, las pruebas de paternidad y los análisis forenses; este último en casos criminales para establecer la relación de un sospechoso con la evidencia dejada en la escena de un crimen (p. ejem. mancha de sangre o semen), para incriminarlo o, en su caso, exonerarlo. Otras aplicaciones menos comunes son para establecer relaciones familiares de restos cadavéricos en casos de desastres o personas desaparecidas, en investigaciones históricas (p. ejem. restos de la familia Romanov, origen de Cristobal Colón, etc.), o en antropología al analizar poblaciones humanas para estudiar su origen y evolución. Esta revisión sobre la prueba de ADN se centrará en su aplicación en paternidad, revisando para ello aspectos básicos del genoma humano y de los marcadores moleculares, técnicas para obtener un perfil de ADN, aspectos bioestadísticos para la interpretación del resultado emitido por un laboratorio, y preguntas frecuentes sobre la prueba de ADN.
 |
Figura 1.
Alec Jeffreys, quién ideo el uso del ADN para identificación humana en Leicester, UK. Al fondo se observa la huella genética del ADN (DNA fingerprinting) como Jeffreys llamo al patrón de bandas que obtuvo, parecido a un código de barras, presumiblemente único para cada individuo. |
Genoma Humano
La estructura del ácido desoxirribonucleico o ADN es bien conocida: una doble cadena que gira sobre sí que contiene información hereditaria codificada solo por cuatro “letras” o nucleótidos[4]: A, G, C y T (adenina, guanina, citosina y timina, respectivamente) (Figura 2). El ADN es importante porque su información es necesaria para el funcionamiento de la célula, ya que contiene las “instrucciones” para sintetizar proteínas[5], quienes propiamente llevan a cabo las diferentes funciones celulares, en forma de enzimas, hormonas, receptores, transportadores, moléculas estructurales, de contracción, soporte, inmunológicas, etc.
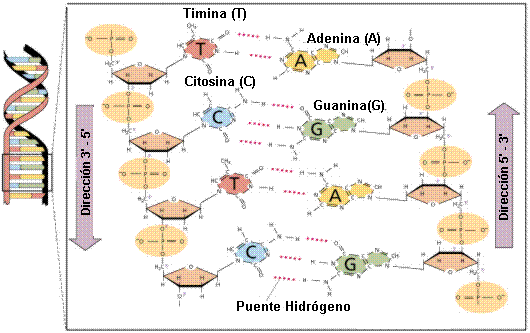 |
Figura 2.
Estructura de doble hélice (izquierda) del ADN o ácido desoxirribonucleico. Los nucleótidos (A, G, C, y T) se encuentran en el interior de la doble cadena de ADN. |
A la dotación completa de material genético que recibimos de nuestros padres se le denomina genoma[6], y se localiza en el núcleo de prácticamente todas nuestras células, por lo que teóricamente es posible realizar la prueba de ADN a partir una sola célula de casi cualquier tejido. Los humanos recibimos una dotación genética doble, vía paterna y materna, que constituye nuestro genoma, y que contiene 6,000 millones de nucleótidos. De toda la información que constituye el genoma, solo una pequeña parte sirve o se “expresa” para formar proteínas (menos del 5%); a los fragmentos del genoma con una secuencia de nucleótidos que sirve para formar una proteína se les denomina genes[7] (Figura 3).
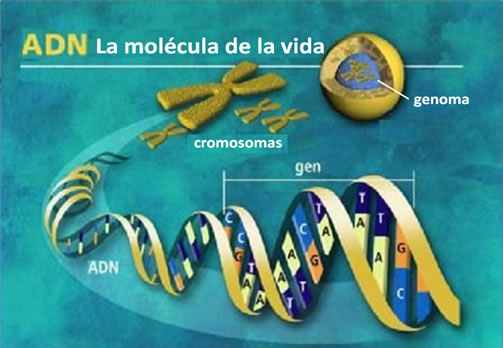 |
Figura 3.
Localización del genoma humano en el núcleo de la célula. Se muestra un cromosoma y un gen, este último caracterizado por tener información para sintetizar una proteína |
Marcadores Moleculares
Considerando la gran cantidad de información que contiene el genoma, podemos visualizar su potencial para identificación humana. En particular la prueba de ADN se realiza analizando secuencias del genoma muy variables, es decir, que entre los individuos de una población puede tener diferentes formas alternas denominadas alelos . Estas secuencias permiten diferenciar a un individuo de otro y, al heredarse de padres a hijos, también permite establecer relaciones biológicas de parentesco, por lo que se les conoce como marcadores genéticos o marcadores moleculares (por estar en la molécula del ADN). Cabe señalar que para cada marcador una persona tendrá dos alelos, uno materno y otro paterno, y a dicha combinación de alelos que recibimos de nuestros padres se le denomina genotipo . Específicamente las secuencias o marcadores empleados para realizar una prueba de ADN se caracterizan por tener repeticiones cortas en tándem , y son ampliamente conocidos por sus siglas en inglés como STRs (short tandem repeats) o microsatélites. A lo largo del genoma se encuentran miles de STRs que pueden ser usados como marcadores moleculares. Como su nombre lo dice, los STRs se componen de secuencias cortas repetidas, por ejemplo GATA, formando diversos alelos que se nombran por el número de veces que se encuentre la secuencia repetida; por ejemplo, el alelo 6 presentará seis veces la secuencia (p. ejem. GATA,GATA,GATA,GATA,GATA,GATA), y en una población podrían existir los alelos 6, 7, 8, 9, 10, 11, 12, 13, 14, etc. El par de alelos o genotipo de una persona para cada marcador STR (p. ejem. 6/9), permite diferenciarlo o relacionarlo con otras personas (Figura 4). Cuando la persona presenta dos alelos diferentes (uno materno y otro paterno), se dice que su genotipo es heterocigoto ; mientras cuando tiene un solo alelo se asume que recibió el mismo alelo de ambos padres, y se dice que su genotipo es homocigoto para el STR en cuestión. En una pareja de heterocigotos para alelos diferentes se pueden generar cuatro genotipos distintos en sus hijos, lo que permite diferenciar individuos estrechamente relacionados como los hermanos (Figura 4). Un perfil de ADN , que también suele llamarse huella genética, se genera obteniendo los genotipos de varios STRs, formando un código que presumiblemente puede llegar a ser único e irrepetible (6/9, 12/16, 17/21, 22/23, etc.). Hay que señalar que la nomenclatura (rango de alelos) de cada STR puede ser diferente, por ejemplo, para el marcador X los alelos pueden ir del 9 al 12, mientras para el marcador Y la nomenclatura va del 12 al 33, etc.
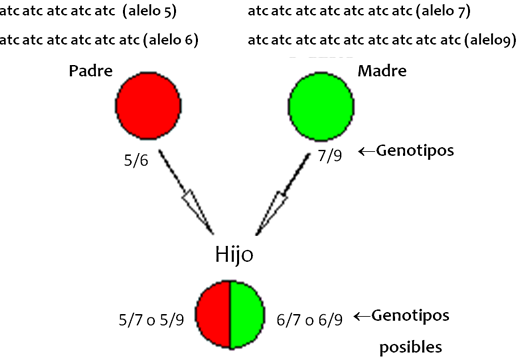 |
Figura 4.
Uso de los marcadores STRs para identificación humana. Los genotipos permiten establecer las relaciones de parentesco y diferenciar a un individuo de otro. De una pareja de heterocigotos se generan multiples genotipos en sus hijos. |
Técnica para obtener el Perfil de ADN
La forma de obtener un perfil de ADN se basa en generar millones de copias o amplificar las secuencias del genoma que permiten diferenciar individuos, en este caso los marcadores STRs. Esta técnica permite replicar al ADN in vitro, y se conoce ampliamente por sus siglas en inglés como PCR[15] (polymerase chain reaction). La PCR se realiza por ciclos de temperatura en los que básicamente suceden los siguientes tres pasos en cada ciclo: 1) desnaturalización, por calor se abren las cadenas de ADN, 2) alineamiento de secuencias cortas conocidas como primers[16], que delimitan las regiones que se van a replicar, y 3) extensión, formándose cadenas nuevas de ADN por acción de la Taq polimerasa[17], una enzima termoestable capaz de añadir nucleótidos a los extremos de los primers. En cada ciclo de PCR se duplica la secuencia de interés (STRs), por lo que en solo 25 ciclos teóricamente habrá millones de copias, a lo que se conoce como “amplificado”, lo que facilitará enormemente su análisis posterior (Figura 5). Para el análisis post-PCR hay que recordar que los alelos STRs se diferencian por el número de veces que se repite una secuencia, esto significa que el tamaño o longitud va a ser diferente de un alelo de otro (Figura 4). Para ver estas diferencias se emplea la electroforesis[18], técnica para separar moléculas en una superficie de soporte (gel) por acción de un campo eléctrico en base a la carga y tamaño de la molécula; las más pequeñas corren más rápido mientras las grandes se van retrasando (Figura 6).
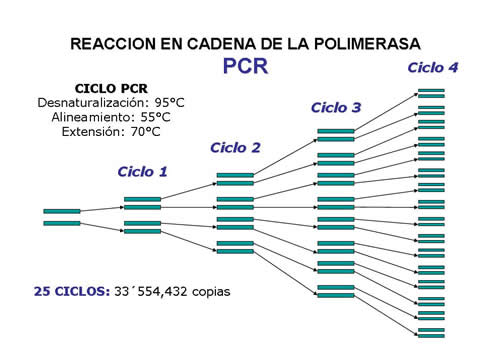 |
Figura 5.
Amplificación exponencial de una secuencia de ADN por ciclos de temperatura por la técnica de PCR |
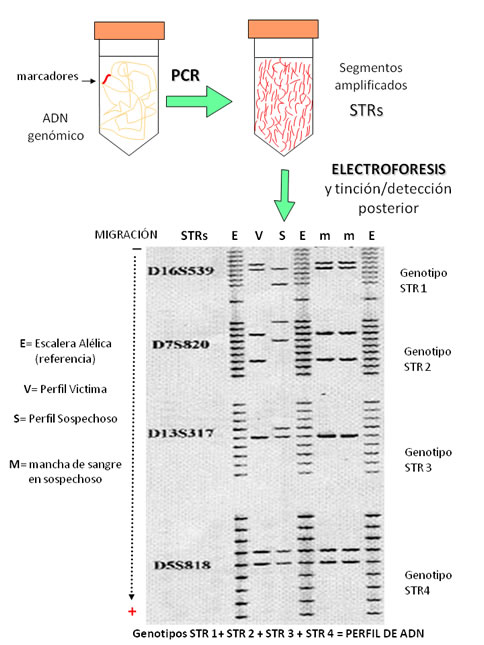 |
Figura 6.
Amplificación de STRs y detección por electroforesis en geles verticales de poliacrilamida en un caso forense. Nótese que el perfil de ADN de la mancha de sangre en ropa del sospechoso es igual al de la víctima. |
Para ello simultáneamente se someten a electroforesis muestras con fragmentos de tamaño conocido, también llamados marcadores de peso molecular o estándar de tamaño, y/o una mezcla de los diferentes alelos posibles para los STRs que se están analizando, y que se conoce como escalera alélica[19] o ladder; con lo que resulta relativamente sencillo definir los alelos/genotipos y posteriormente el perfil genético de una persona. La electroforesis en una prueba de ADN se realiza de dos formas: 1) por geles verticales de poliacrilamida, técnica que ha caído francamente en desuso, y 2) electroforesis capilar (EC)[20], cuyo proceso es más preciso y automatizado, por lo que constituye el método de elección en los laboratorios de genética forense en el mundo. Finalmente, para observar el ADN amplificado (STRs) y sometido a electroforesis es necesario teñirlo; existen métodos tradicionales como la tinción con nitrato de plata de los geles de poliacrilamida (Figura 6), o más sofisticados que involucran la detección de fluorescencia añadida durante la PCR y que se detecta en sistemas automatizados de electroforesis capilar.
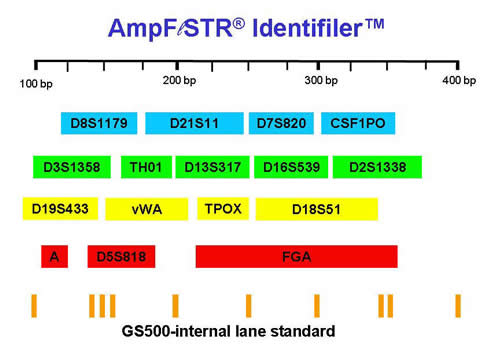
La siguiente descripción se enfocará en el análisis de varios STRs simultáneamente (PCR múltiplex), seguido por electroforesis capilar (EC) para obtener un perfil de ADN, donde existen diferentes kits o sistemas genéticos[21] que permiten amplificar hasta 16 marcadores, 15 STRs y un marcador sexual llamado amelogenina[22] que define el sexo de la muestra. Los kits comerciales más empleados son AmpFlSTR® Identifiler™ kit (Applied Biosystems, Foster City, CA), y el PowerPlex 16 (Promega Corp., Madison, CA). En ambos casos, durante la PCR los productos amplificados (STRs) se marcan con diferentes fluorocromos[23], formándose así grupos de STRs que fueron marcados con el mismo color, pero evitando que alelos de STRs diferentes se sobrelapen en tamaño (Figura 7).
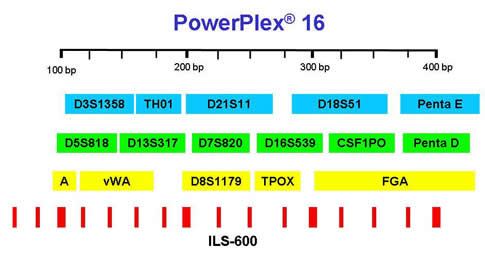 |
Figura 7.
Los dos principales kits comerciales para identidad humana que analizan 15 STRs y el marcador sexual amelogenina (A). Los nombres de los STRs están en recuadros de colores, y en la parte de abajo un estándar de tamaño o marcador de peso molecular para definir el tamaño de los alelos STRs. Nótese que cada STR abarca un rango de tamaño por los diferentes tamaños que sus alelos pueden tener, y que no se sobrelapa con otro STR. |
 |
Los amplificados deben someterse a electroforesis capilar, es decir, este proceso se hace a través de un tubo del tamaño de un capilar relleno de un polímero, donde bajo el mismo principio de separación por la carga y tamaño de la molécula, los amplificados se van separando al aplicar corriente eléctrica. Para este fin se usan analizadores genéticos, como el ABI-Prism 310 de Applied Biosystems, que de forma automatizada carga la muestra en el capilar para luego aplicar voltaje. De esta forma se separan los STRs hasta llegar a una ventana donde pasa un rayo laser que excita a los fluorocromos, que a su vez emiten fluorescencia detectada en una cámara CCD, la cual convierte esa luz en impulsos electrónicos que, con ayuda de un software, se representan gráficamente en un electroferograma[24] (Figura 8).
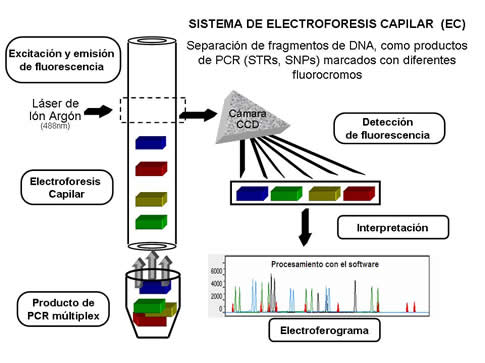 |
Figura 8.
Representación de los pasos que involucra el análisis de fragmentos de ADN por electroforesis capilar (EC). |
Cabe señalar que la luz (longitud de onda) de las diferentes fluorescencias se sobrelapa, y la separación de colores se logra mediante el uso de valores que indican cuanto se sobrelapan cada uno de los colores, a lo que se denomina matriz. Durante el análisis del electroferograma para definir el perfil de ADN es muy importante que simultáneamente se corra en la EC una escalera alélica (ladder) con todos los alelos los STRs, lo que por comparación directa se facilita enormemente la asignación correcta de los alelos, genotipos, y finalmente perfil de ADN del individuo. Cabe señalar que todo este proceso dura alrededor de 30 minutos por muestra, facilitando enormemente esta labor. Una vez realizada la electroforesis capilar se observa el perfil de ADN de un individuo en un electroferograma, donde los picos de acuerdo a su color y posición nos indican los alelos (genotipo) que tiene la persona para cada STR. Si un individuo presenta uno o dos picos (alelos), indica que su genotipo es homocigoto o heterocigoto, respectivamente, para el STR en cuestión (Figura 9).
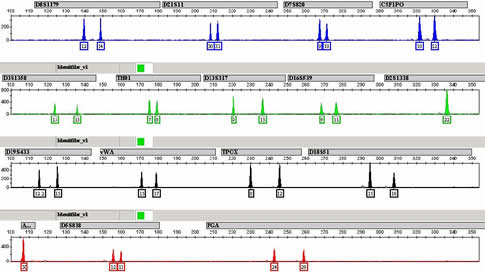 |
Figura 9.
Electroferograma del perfil genético de un individuo. El tamaño en nucleótidos (pb) y nombre del STR se muestra en la parte superior de cada carril. Debajo de cada pico se indican los alelos del individuo. Para amelogenina (A) se observa solo un pico y una X (mujer, XX). |
Interpretación de resultados
Después de obtener los perfiles en una prueba de ADN para determinar la paternidad entre un supuesto padre y un hijo en disputa, existen dos resultados posibles al comparar sus perfiles: 1) Que para al menos dos marcadores no exista ninguna coincidencia entre los alelos/genotipos del supuesto padre e hijo, lo que se denomina exclusión[25], y se descarta de inmediato la paternidad biológica (Figura 10); 2) Que sí coincida el padre con el hijo en al menos un alelo en todos los marcadores analizados, lo que se puede interpretar como que el supuesto padre es el padre biológico del hijo en disputa. Pero cuidado, antes de llegar a esta conclusión se debe hacer una valoración bioestadística del caso que permita contestar la siguiente pregunta: ¿qué tan probable sería que el perfil de ADN de cualquier persona, que NO sea el padre biológico, hubiera coincidido por azar o casualidad con el perfil de ADN del hijo en disputa? (Figura 10). Para hacer esta valoración correctamente es necesario saber la frecuencia en la población de los alelos que concuerdan entre el supuesto padre e hijo. Para entender la importancia de estos datos poblacionales en la interpretación de una prueba de paternidad vamos a poner un ejemplo. Supongamos que en una prueba de paternidad el alelo concordante entre el supuesto padre e hijo lo tiene el 95% de las personas de la población mexicana, ¿esta sería una prueba contundente en el caso?, seguro que no, inclusive podemos inferir que esta es una evidencia muy pobre y que sería injusto concluir, considerando solo esta evidencia, que el supuesto padre es realmente el padre biológico, ya que la mayoría de varones que hubieran puesto en su lugar hubiera coincidido con el hijo. Por el contrario, si el alelo concordante entre el supuesto padre e hijo es muy raro y se observa solo en 1 de 10,000 personas de la población, intuitivamente inferimos que esta evidencia es valiosa para establecer la paternidad, ya que sería muy poco probable que hubiera concordado el hijo con el supuesto padre si éste último no lo fuera realmente.
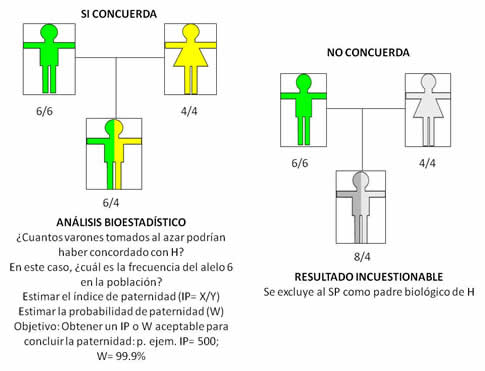 |
Figura 10.
Diferencia en la interpretación de un análisis de paternidad donde concuerda el supuesto padre (SP) y el hijo (H), respecto a una exclusión de paternidad. |
Las frecuencias poblacionales para hacer esta interpretación se obtienen de estudios en la población donde se realizan las pruebas de ADN y, preferentemente, deben estar publicados en revistas científicas serias (indizadas). En México ya se cuenta con varios estudios que permiten hacer esta valoración y la mayoría se citan al final de este trabajo. Se abordará la explicación sobre cómo interpretar una prueba de paternidad biológica con ADN a partir de un ejemplo donde la prueba indique paternidad, discutiendo posibles cuestiones que pudieran surgir al público no-experto.
Índice de Paternidad (IP) y Probabilidad de Paternidad (W)
Haciendo algunas simplificaciones diremos que la interpretación de un caso de paternidad consiste en contrastar dos hipótesis o posibilidades contrarias:
X: el supuesto padre (SP) es el padre biológico del hijo
Y: el supuesto padre (SP) NO es el padre biológico y por lo tanto es otro hombre
A este contraste se le conoce como índice de paternidad (IP), que indica cuantas veces es más probable que el SP sea el padre (X), respecto a que no lo sea (Y), y suele describirse como: IP = X/Y.
El IP se estima a partir de los alelos/genotipos de cada STR usado en una prueba de paternidad; luego se obtiene un IP total multiplicando el IP de todos los STRs, es decir: IP total= IPSTR-1 x IPSTR-2 x IPSTR-3 …etc. Otra manera de interpretar el IP total es convirtiéndolo a probabilidad de paternidad, ampliamente conocido como W, usando la siguiente fórmula:
W = IP/ (1+ IP)
Ejemplo 1 (trío)
Ahora vamos a emplear las fórmulas en casos reales a partir de diferentes combinaciones de genotipos. Comenzaremos con un caso de un trío donde participan padre, madre e hijo (Cuadro 1).
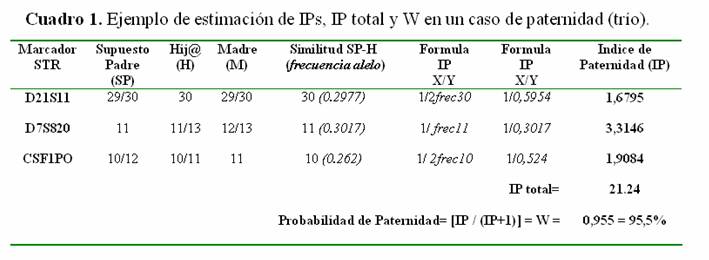
De la formula de IP= X/Y, normalmente X equivale en probabilidad a uno (1), ya que asume que el SP sí es el padre biológico; otra forma de describir esta probabilidad de haber encontrado esa concordancia pero en porcentaje es 100%. Mientras como Y asume que SP no es el padre, entonces se infiere que fue otra persona de la población y su probabilidad corresponde a la frecuencia poblacional del alelo concordante entre SP y H. Estas frecuencias se obtienen preferentemente de estudios genético-poblacionales del lugar donde se hizo la prueba, ya que las frecuencias pueden variar entre una población y otra. Otro detalle importante que se observa en las fórmulas para calcular el índice de paternidad (IP) del Cuadro 1, es que no todas son iguales, principalmente cambia la probabilidad de que sea un hombre al azar (Y), ya que la fórmula depende de la combinación de genotipos del trío, y particularmente involucra al alelo que concuerda entre SP y H. Por ejemplo, la lógica de la fórmula empleada en el los genotipos del trío para el primer STR (D21S11), es que el hijo es homocigoto 30 (30/30) por lo que hay dos posibilidades de que el hij@ haya recibido al alelo 30 de una hombre al azar de la población, ya sea de parte de la madre o del padre, de allí que resulte la fórmula igual a 1/frec (frecuencia del alelo concordante SP-H). Explicar los detalles de cada una de las formulas para los posibles genotipos de un trío se escapa de los objetivos de esta revisión, por lo cual se recomienda la lectura el libro de Evett y Weir (1998).
Ejemplo 2 (dúo)
Este caso es relativamente común y quién no participa normalmente es la madre. Se tomará la misma combinación de genotipos del ejemplo anterior eliminando a la madre, para notar los cambios en las fórmulas y el resultado final (Cuadro 2).
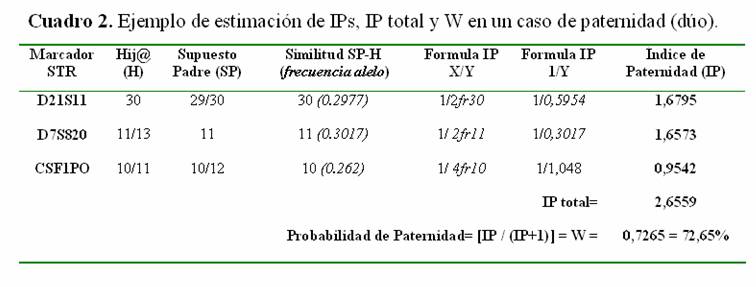
Comparando el cuadro l y 2 inmediatamente sobresalen dos aspectos: 1) Cambian algunas formulas, y por lo general disminuye la probabilidad de paternidad (W) y los índices de paternidad (IPs) estimados. Esta disminución se debe a la información que se pierde de la madre, por lo que no se puede estar seguro cuál de los alelos del hijo es el paterno y cuál es el materno (i. e. 10/11 para CSF1PO), mientras en el caso anterior se podía inferir fácilmente (11 para CSF1PO). Por lo anterior, al estimar que cualquier otro hombre de la población sea el padre biológico de H, se amplían las posibilidades. A pesar de esto, en general los sistemas genéticos comerciales que incluyen 15 STRs logran valores de W> 99.9%. Existen otras situaciones de paternidad más complejas donde cambia la interpretación matemática o bioestadística, como 1) establecer una paternidad cuando se duda de ambos padres, por ejemplo cuando se piensa que se les cambio al bebé en el hospital o se quiere emparentar restos cadavéricos con una pareja de supuestos padres, 2) cuando no está el padre y se quiere saber si dos individuos son hermanos, 3) cuando no está el padre pero se cuenta con los abuelo paternos, etc. La interpretación bioestadística en cada caso tiene que analizarse por separado para llegar a una conclusión científicamente correcta, e igual se recomienda a los interesados la lectura del libro del libro de Evett y Weir (1998).
¿Cuántos marcadores son necesarios para la prueba?
Se ha sugerido por parte del FBI[26], entre otros organismos, el uso de 13 STRs para identificación genética humana que constituyen el CODIS[27] (Figura 11), los cuales son suficientes para resolver la mayoría de casos de paternidad. Sin embargo, en realidad el número de marcadores depende del caso, pudiendo ser menor o mayor a 13 marcadores; en realidad lo fundamental es llegar a una conclusión lo suficientemente confiable. ¿Cuándo es suficientemente confiable?, esta pregunta tiene una respuesta en realidad subjetiva que suele ser arbitraria. Hasta hace algunos años se empleaban algunos enunciados para interpretar el porcentaje de paternidad (W); entre los más famosos están los postulados de Hummel, donde W≥ 99,73% (equivalente a un IP de 400) se traducía como “paternidad prácticamente probada” y era límite máximo para valorar W. El desarrollo científico tecnológico hace que estos enunciados y valores a la fecha se consideren obsoletos e insuficientes. Actualmente entre los laboratorios hay dos tendencias: 1) reportar el valor de W e IP obtenido a partir del sistema genético ofertado; o 2) fijar un límite mínimo, por ejemplo de 1,000 o hasta 10,000 para el IP. En caso de no llegar al límite mínimo establecido se procedería a incrementar el número de marcadores, hasta alcanzar una exclusión clara o un valor aceptable de IP.
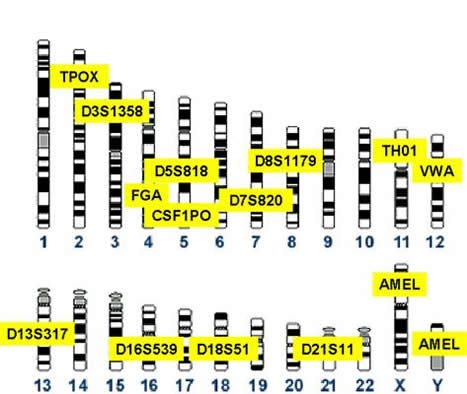 |
Figura 11.
Nombres de los 13 marcadores STRs que constituyen el Combined DNA Index System (CODIS), así como su posición en los cromosomas del genoma humano. |
Exclusiones
En un caso estándar de un trío de SP-H-M analizado con 13 a 16 marcadores, es típico encontrar de 7 a 10 exclusiones, lo que no significa que podamos encontrar un caso con sólo 2 o 3 exclusiones, u otros con 12. En casos de paternidad con un solo progenitor el número de exclusiones típico es entre 4 y 5. En casos más complicados donde se pierde información genética se reduce el número de exclusiones que se espera encontrar, por lo que hay que aumentar el número de marcadores investigados. Existe un caso particular denominado exclusión de segundo orden[28], donde SP y H son homocigotos para alelos diferentes. Este resultado se puede explicar, además de la no-paternidad, porque comparten un alelo “invisible” (alelo nulo) y SP realmente es el padre biológico del hijo en disputa. Todos los demás casos de exclusiones se denominan de primer orden.
Oídos sordos a los IPs cuando se establece la exclusión
¿Qué se debe hacer cuando en una prueba con 13 marcadores, 3 excluyen o indican que “no es el padre” y los otros 10 concuerdan entre SP y H, lo que alguien podría interpretar como indicio de paternidad por ser la mayoría?, ¿A quién le hago caso?, ¿Cómo lo interpreto?. La respuesta es: Cuando se ha llegado a la conclusión de exclusión, por haber encontrado al menos dos exclusiones de primer orden, se hace oídos sordos hacia los demás marcadores. ¿Cómo se explica esto?. Sucede que los alelos detectados con marcadores en una prueba de ADN no son exclusivos de una familia, en realidad en una población mucha gente tiene los mismos alelos o genotipos sin que esto signifique que estén emparentados; es como tener ojos cafés, pelo negro o castaño en la población mexicana, mucha gente presenta estos rasgos sin que sean familiares cercanos. La fortaleza de la prueba es que se generan muchas combinaciones distintas entre los individuos de la población, aún entre hermanos, por lo que es muy poco probable que al tomar dos individuos al azar ellos tengan ya sea el mismo perfil de ADN (caso forense), o que un varón concuerde con un hijo en disputa, en al menos un alelo por cada STR de sus perfiles genéticos (caso de paternidad). Clásicamente con más de una exclusión de primer orden se consideraba una exclusión probada (definitivamente no es el padre), aunque cada vez se tiene más cuidado porque se han descrito casos de paternidad con hasta 3 cambios en el material genético (mutaciones) que ocasionan que no coincidan el SP y H[29], aunque el SP si sea padre biológico de H.
Como interpretar las mutaciones en paternidad
En un caso típico de mutación, una prueba de ADN con 13 o 15 marcadores indicaría que solo uno de ellos no concuerda entre SP y H, lo que hace pensar que la única exclusión observada en realidad se trata de una mutación. Una de las soluciones más sencillas se basa en la sugerida por la AABB[30], donde el IP del marcador que indica exclusión se obtiene con una formula sencilla IP= µ/PE. El símbolo mu (µ) indica la tasa de mutación que se ha estimado en general para los marcadores STRs (i. e. 7x10-3), y el poder de exclusión (PE)[31], parámetro de paternidad estimado y reportado junto con las frecuencias alélicas en los estudios poblacionales antes mencionados.
¿Paternidad sin el padre? Uso del cromosoma sexual Y
Cada vez que la célula se va a dividir el material genético se compacta en estructuras llamadas cromosomas[32]. La presencia del cromosoma Y en el esperma determina el sexo masculino en el futuro hijo en la mitad de las fertilizaciones, y establece una herencia exclusiva del cromosoma Y de padres a hijos varones (Figura 12). A diferencia de la mayor parte del material genético en el humano (23 cromosomas maternos y 23 paternos), los cromosomas Y no se “mezclan” durante la formación de los gametos en la meiosis[33]. Por esta razón, cada hombre recibe un cromosoma Y idéntico al de su padre. Cabe mencionar que existe una pequeña región en los extremos del cromosoma Y (aprox. 5%) que sí se “mezcla” con el otro cromosoma sexual (X). Dicha región en general es de poco interés con fines de identificación humana.
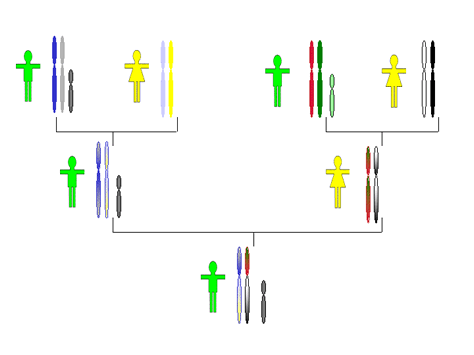 |
Figura 12.
Comparación de la herencia directa varón-varón del cromosoma Y (gris pequeño). Los demás cromosomas se “mezclan” cuando pasan de una generación a la siguiente. Por esta razón, los varones tenemos un cromosoma Y prácticamente igual al de nuestros padres y abuelos paternos. |
Cuando se analizan varios marcadores del cromosoma Y, estos se heredan en bloque o en conjunto de padres a hijos varones, a lo que se le conoce como haplotipo[34]. También para el cromosoma Y los marcadores más utilizados en identificación humana son los STRs, ya que presentan un mayor número de alelos en la población, y a su vez una mayor capacidad de discriminar dos haplotipos y/o varones no-relacionados. Al analizar varios STRs del cromosoma Y (Y-STRs) en un varón se forma un código que constituye su perfil de ADN masculino. El análisis de laboratorio de los Y-STRs es el mismo que para los STRs no sexuales usados en las pruebas de paternidad “normales”, es decir PCR múltiplex y electroforesis capilar. Existen kits comerciales para este análisis, donde destacan los kits Powerplex 16 (Promega Corp.) y el Y-filer (Applied Biosystems) y donde se analizan 12 y 16 Y-STRs, respectivamente (Figura 13).
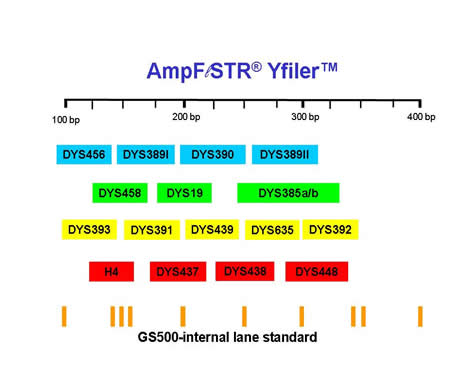 |
Figura 13.
Kit comercial para analizar 16 Y-STRs del cromosoma Y. Se muestran los colores (fluorocromos) con los que están marcados los diferentes Y-STRs. Se describe el rango de tamaño de los diferentes alelos de cada Y-STR en pares de bases (pb). |
En casos criminales o análisis forense, el potencial del cromosoma Y recae en el hecho de que la mayoría de crímenes violentos son llevados a cabo por varones. Además, permite resolver casos en situaciones forenses particulares, como violaciones, donde existe una mezcla de ADN de hombre/mujer, ya que estos marcadores al ser específicos del varón generarán un perfil de ADN específico del agresor. Por su parte, en pruebas de paternidad, los Y-STRs tienen un gran poder de exclusión cuando el hijo en disputa es varón, y permiten resolver casos donde el supuesto padre no está disponible, ya que sus parientes varones por línea paterna (hermanos, tíos, abuelo, primos, etc.) sirven de referencia por tener el mismo haplotipo para el cromosoma Y (Figura 14).
 |
Figura 14
Establecimiento de paternidad mediante Y-STRs en un caso donde el supuesto padre no está disponible. Se toma como referencia cualquier varón emparentado vía paterna. La exclusión es irrefutable, pero la concordancia debe confirmarse con STRs no sexuales. |
Es importante considerar que los haplotipos del cromosoma Y son compartidos por todos los parientes vía paterna, que constituyen los llamados linajes paternos y que suelen agruparse dentro de las poblaciones de formas muy particulares de acuerdo a sus historias. Por consiguiente, es necesario considerar que en una prueba forense o de paternidad puede existir un número indeterminado de varones con el mismo haplotipo, además de los parientes por línea paterna del sujeto implicado. Por esta razón, aunque los haplotipos Y-STRs tienen un gran poder de exclusión y esta no necesita ser confirmada, en casos donde concuerda el SP-H o el sospechoso con la evidencia del crimen, es conveniente confirmar la relación biológica con STRs no-sexuales. La desventaja más importante del cromosoma Y en pruebas de paternidad es que sólo sirve en aproximadamente el 50% de los casos, es decir, cuando el hijo en disputa es varón.
Interpretación de haplotipos del cromosoma Y en identidad humana
A diferencia de las pruebas de ADN normales donde se considera la frecuencia de cada uno de los alelos que concuerdan entre SP y H, para el cromosoma Y que se hereda en bloque, se debe considerar la frecuencia de toda la combinación de alelos, es decir, el haplotipo completo. En este sentido, los estudios en Mexico son más escasos que los STRs no-sexuales, aunque recientemente hemos publicado la base de datos de Y-STRs que casi triplicará las poblaciones hasta ahora estudiadas (Salazar-Flores y cols. 2009). La forma más sencilla de comparar la frecuencia de un haplotipo de Y-STRs en un caso de identidad humana es vía internet, ya que los datos antes mencionados se han ingresado a una base de datos internacional (www.yhrd.org). En esta página se puede ingresar un haplotipo de Y-STRs para que se busque cuantas veces se ha observado en alguna población seleccionada o en toda la base de datos, y con esta información se procede a estimar el índice de paternidad (o en su caso de hermandad vía paterna) así como la probabilidad respectiva.
Ejercicios de Control de Calidad
El más reconocido por su trayectoria desde 1992 e impacto en el ámbito científico de poblaciones hispanoamericanas es el GEP-ISFG (www.gep-isfg.org) o Grupo Español-Portugués de la Sociedad Internacional de Genética Forense (International Society of Forensic Genetics), que organiza ejercicios de colaboración con el objetivo de avanzar en la estandarización de los métodos empleados por distintos laboratorios para el análisis de marcadores genéticos para identificación humana. El ejercicio de control es coordinado por la Unidad de Garantía de Calidad del Departamento de Madrid del Instituto Nacional de Toxicología. En este ejercicio pueden participar todos los laboratorios interesados del grupo. A cada laboratorio se le asigna un código para garantizar el anonimato de los resultados. A los laboratorios participantes se les remiten seis manchas (preferentemente de sangre, pudiendo incluir otro fluido biológico) sobre distintos soportes, se les solicita la obtención de perfiles de ADN con los marcadores habitualmente utilizados en su laboratorio. En el ejercicio se incluye una investigación de paternidad, solicitando a los laboratorios una valoración estadística de resultados y una interpretación final. Los resultados se recogen sobre un formulario vía internet que incluye detalles sobre metodología y resultados obtenidos los cuales deben ser enviados por el laboratorio en un periodo de tiempo de tres meses desde la recepción de las muestras. Se ofrece la posibilidad al laboratorio de enviar resultados para cada marcador al control de calidad o simplemente como intercambio de resultados para aquellos nuevos marcadores, en los que se está montando y validando la técnica. El centro coordinador elabora un informe con el resumen de la información remitida por los laboratorios, incluyendo metodologías y resultados. El centro coordinador emite certificados para los laboratorios participantes, incluyendo los marcadores en los que ha participado y obtenido un resultado acorde con el consensuado y en los que hay al menos tres laboratorios que han emitido resultados.
Otra de las instancias que organiza ejercicios de control de calidad es el SLAGF o Sociedad Latinoamericana de Genética Forense (www.slagf.org). La mecánica es similar, se reciben muestras en papel, se procesan para obtener el perfil genético con los sistemas genéticos del laboratorio, se reportan los perfiles en un formulario y además de resuelve un caso teórico de paternidad. Si se ha resuelto adecuadamente el ejercicio se emite una constancia.
Conclusiones
En los últimos años el desarrollo de las técnicas de biología molecular ha facilitado enormemente la tarea de identificación genética, gracias a sistemas genéticos estandarizados (STRs) que pueden compararse entre laboratorios. A pesar del desarrollo de nuevas tecnologías para analizar marcadores genéticos de un nucleótido (SNPs[35]), suena difícil que desplacen a los STRs de las cuales se han generado ya bases de datos gigantescas en diferentes países, principalmente como auxiliares en la investigación criminal, por ejemplo comparando el perfil de ADN de una escena de un crimen con una base de datos que facilita la labor de buscar sospechosos.
Sin embargo, cabe señalar la importancia de la capacitación de personal de laboratorios y los involucrados en la impartición de justicia, ya que desde mi punto de vista este es uno de los retos más importantes en México para lograr la correcta interpretación de la prueba de ADN con bases científicas, estableciendo perfectamente la responsabilidad tanto de los peritos en genética forense como de los jueces. Por ejemplo, en un caso criminal la función del perito no es señalar si los sospechosos son o no culpables, sino establecer si, por ejemplo, el perfil de ADN de una mancha de sangre encontrada en la escena del crimen corresponde a la del sospechoso, lo que indica que éste estuvo allí más no que él sea un criminal. En cambio, al juez le tocará decir la culpabilidad o inocencia del sospechoso, considerando no solo la evidencia genética, sino también la información no genética de relevancia, por ejemplo si conocía o había amenazado a la víctima, si tiene antecedentes de violencia, si lo vieron en la zona antes del crimen, etc. En paternidad, aunque el resultado de la prueba sí se relaciona directamente con la resolución que dará finalmente el juez, debe considerarse la posibilidad de situaciones especiales como que el supuesto padre haya recibido un transplante (lo que cambiaría su perfil de ADN), o que el padre biológico estuviera relacionado con él supuesto padre, lo que incrementaría la posibilidad de coincidencia. Finalmente, esto nos llevar a concluir que las pruebas de ADN actualmente son técnicamente confiables y relativamente fáciles de realizar, nuestro mayor reto ahora es que el público y el personal de laboratorio y de justicia la entienda correctamente, para lo cual se realzó este trabajo.
Literaturas Recomendadas
Butler JM. (2005). Forensic DNA typing. Elsevier Academic Press, Burlington, MA, USA . p 201-240.
Butler JM. (2006) Genetics and Genomics of Core Short Tandem Repeat loci used in Human Identity Testing. Journal Forensic Sciences 51(2): 253-265.
Evett IW, Weir BS (1998). Interpreting DNA evidence. Sinauer. MA, USA .
Jobling MA, Gill P. (2004) Encoded Evidence: DNA in forensic analysis. Nature Reviews (5):739-752.
Ma. Begoña Martínez Jarreta (1999). La prueba del ADN en Medicina Forense., Masson (Ed.), Barcelona, España.
Estudios Poblacionales para la interpretación de la Prueba de ADN en México
Rangel-Villalobos H, Rivas F, Torres-Rodriguez M, et. al (1999). Allele Frequency Distribution of Six Amp-FLPs (D1S80, APO-B, VWA, TH01, CSF1PO and Hprtb) in Mexican Population. Forensic Science International, 105:125-129.
Rangel-Villalobos H, Jaloma-Cruz AR, Sandoval L, Velarde-Felix JS, Gallegos-Arreola MP, Figuera LE (2001). “Y-chromosome Haplotypes for Six STRs is a Mexican Population”. Archives of Medical Research Jun 32 (3): 61-66.
Rangel-Villalobos H, Jaloma-Cruz AR, et al. (2001). “Male DNA profile: Y-chromosome haplotypes in a Mexican population analyzing five STRs”. Revista de Investigación Clínica 53 (5): 401-406
Cerda-Flores RM, Budowle B, Jin L, Barton SA, Deka R, Chakraborty R. (2002). Maximum likelihood estimates of admixture in northeastern Mexico using 13 short tandem repeat loci. Am J Hum Biol 14:429-439.
Luna Vázquez A, Páez-Riberos LA, González-Martín A, Muñoz-Valle F, Rangel-Villalobos H, (2003). “Genetic Distribution of nine STRs in a Mexican Population”. Forensic Science International 136(1-3): 96-98
Hernández-Gutiérrez S, Hernández-Franco, Martínez-Tripp, Ramos-Kuri M, Rangel-Villalobos H (2005). STR Data for 15 Loci in a Population sample from the Central Region of Mexico. Forensic Science International 151(1):97-100.
Martínez-González LJ, Martínez-Espín EM, Fernández-Rosado F, Moguel MA, Entrala C, Álvarez C, Lorente JA, Budowle B. (2005). Mexican population data on fifteen STR loci (Identifiler kit) in a Chihuahua (north central Mexico ) sample. J Forensic Sci 50(1):236-238.
Luna-Vázquez A, Vilchis-Dorantes G, Aguilar-Ruiz MO, Bautista-Rivas A, Rojo-Nava AL, Ríos-Barrios E, Rangel-Villalobos H (2005). Population Data for 15 Loci (Identifiler® Kit) in a sample from the Valley of México. Legal Med Oct 7(5):331-3.
Torres-Rodríguez M, Martínez-Cortes G, Páez-Riberos LA, Sandoval L, Muñoz-Valle JF, Ceballos-Quintal JM, Pinto-Escalante D, Rangel-Villalobos H (2006). Forensic potential of the STR DXYS156 in Mexican Populations: inference of X-linked allele null. Legal Med. 2006 Jan;8(1):52-4.
Licea-Cadena RA, Rizzo-Juárez RA, Muñiz-Lozano E, Páez-Riberos LA, Rangel-Villalobos H (2006). Population Data of Nine STRs of Mexican-Mestizos from Veracruz (Central South-Eastern, Mexico ). Legal Medicine Jul; 8(4):251-2.
Gorostiza A, González-Martín A, Ramírez CL, Sánchez C, Barrot C, Ortega M, Huguet E, Corbella J, Gené M. (2007). Allele frequencies of the 15 AmpF/STR Identifiler loci in the population of Metztitlán (Estado de Hidalgo), Mexico . Forensic Sci Int 166(2-3):230-232.
Gutiérrez-Alarcón AB, Moguel-Torres M, León-Jiménez AK, Cuéllar-Nevárez GE, Rangel-Villalobos H. Allele and Haplotype Distribution for 16 Y-STRs (AmpFlSTR® Y-filer™ kit) in the state of Chihuahua at North Center of Mexico. Legal Med 2007 9(3):154-7.
Luna-Vázquez A, Vilchis-Dorantes G, Aguilar-Ruiz MO, Bautista-Rivas A, Pérez-García A, Orea-Ochoa R, Villanueva-Hernández D, Guerra-Tejeda M, Rangel-Villalobos H. (2008). “Allele and Haplotype frequencies of the PowerPlex® Y system in a Population Sample from Mexico City”. FSI Genetics 2(1) e11-e13.
Rangel-Villalobos H, Muñoz-Valle JF, González-Martín A, Gorostiza A, Magaña MT, Páez-Riberos LA (2008). Genetic admixture, relatedness and structure patterns among Mexican populations revealed by the Y-chromosome. Am J Phys Anthrop. 135(4):448-61.
Juárez-Cedillo T, Zuñiga J, Acuña-Alonzo V, Pérez-Hernández N, Rodríguez-Pérez JM, Barquera R, Gallardo G, Sánchez-Arenas R, García-Peña MC, Granados J, Vargas-Alarcón G. 2008. Genetic admixture and diversity estimations in the Mexican Mestizo population from Mexico City using 15 STR polymorphic markers. Forensic Sci Int Genet (2):e37-e39.
Rubí-Castellanos R, Anaya-Palafox M, Mena-Rojas E, Bautista-España D, Muñoz-Valle JF, Rangel-Villalobos H (2009). Genetic Data of 15 autosomal STRs (Identifiler kit) of Three Mexican Mestizo Population samples from the States of Yucatan (Southeast), Jalisco (West), and Puebla (Center). Forensic Sci Int Genet (3)e71-e73.
Rubi-Castellanos R, Martínez-Cortés G, Muñoz-Valle JF, González-Martín A, Cerda-Flores R, Rangel-Villalobos H (2009). Pre-Hispanic Mesoamerican Demography Approximates the Present-day Ancestry of Mestizos throughout the Territory of México. Am J Phys Anthrop. (aceptado, in press).
Salazar-Flores J, Dondiego-Aldape R, Rubi-Castellanos R, Anaya-Palafox M, Nuño-Arana I, Canseco-Ávila LM, Flores-Flores G, Morales-Vallejo ME, Barajas-Pérez N, Muñoz-Valle JF, Campos-Gutiérrez R, Rangel-Villalobos H. (2009). Paternal European impact on present-day Mexican-Mestizos: Evidence from Y-STR haplotypes. Am J Hum Biol (enviado).
Es autor de varios capítulos en libros y artículos de divulgación científica relacionados con la prueba de ADN y la aplicación de marcadores moleculares para estudios en la población mexicana, los cuales están publicados en artículos científicos. Ha dirigido y/o asesorado varias tesis de licenciatura, maestría y doctorado en el área de Ciencias de la Salud, y ha participado como ponente o conferencista en docenas de eventos científicos relacionados con la genética desde 1997. Ha recibido varias distinciones, entre las que destacan: mención honorífica en tesis de maestría (1998), presea al mérito académico en el área de Ciencia e Investigación por el STAUdeG (2002), dos veces 1er lugar y una vez segundo del premio “HECTOR MARQUEZ MONTER” al mejor trabajo de investigación por la Asociación Mexicana de Genética Humana (1999, 2004 y 2008), nombrado “Biólogo Destacado” por los Biólogos Colegiados de Jalisco (2005). Fue asesor del trabajo que obtuvo el “Premio Chihuahua 2006” en Ciencias Biológicas, y ganador de un Premio Nacional del INAH (2007) por su trabajo de divulgación inédito en Antropología Molecular.
Recomendaciones y quejas sobre la atención y servicios ofrecidos por DNA Profile SC son bien recibidos en el correo:

